Prof. Dr. Salih Sanlioglu Akdeniz Üniversitesi Gen ve Hücre Tedavi
advertisement
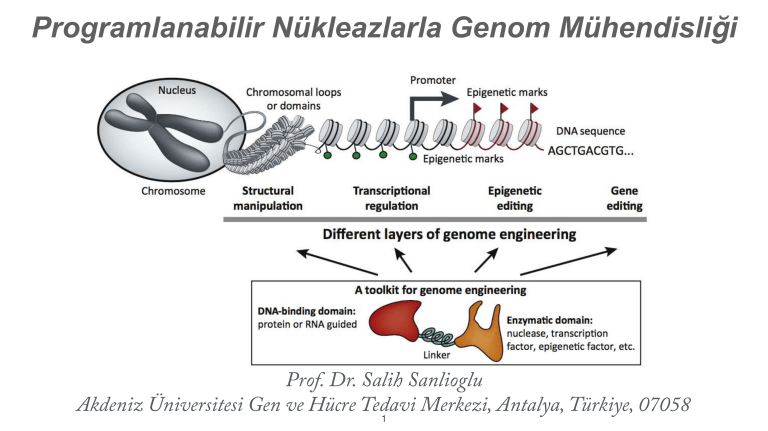
Programlanabilir Nükleazlarla Genom Mühendisliği Prof. Dr. Salih Sanlioglu Akdeniz Üniversitesi Gen ve Hücre Tedavi Merkezi, Antalya, Türkiye, 07058 1 Genom Modifikasyonu: Gerekçe • İnsan genom araştırmalarının temel amacı genlerin fonksiyonlarını ve işlevliliklerini bütünüyle açığa çıkarmaktır. Bir insan hücresinin çekirdeğinde yaklaşık 1 metre uzunluğunda linear bir DNA ve bunun üzerinde de 20,000’den fazla protein kodlayan ve kodlamayan genler vardır. • Genetik hastalıklara karşı etkin terapötik ilaçların geliştirilmesi genlerin fonksiyonlarının tam manasıyla anlaşılmasına bağlıdır. • Genetik değişiklikler ve hastalıklar arasındaki ilişki yıllardır bilinmesine rağmen bazı hastalık fenotiplerinin ortaya çıkmasına sebebiyet veren mutasyonlar ancak genom değişikliği yapılarak giderilebilir ve böylelikle hastalıklar tedavi edilebilir. • Ancak genom karmaşık bir yapıya sahip olduğundan genetik bilgiyi manipüle etmek oldukça zordur. Bu açıdan girişimsel düzeyde genom modifikasyonu teknolojik olarak ileri düzeyde geliştirilmiş moleküler genetik yöntemlerle sağlanabilir. 2 Genom Modifikasyonu Başlangıç • 1988 yılında Rudin ve Haber mayalarda çift zincirli DNA kırıkları oluşturarak (DSBs) gen hedeflemenin (gene targeting) etkinliğinin arttırılabileceğini iddia ettiler. 1994 yılında Jasin’in araştırma grubu memeli hücrelerinde bu durumu doğruladılar. Bu amaçla saccharomyces cerevisiae’den elde edilen I-SceI isimli restriksiyon enziminin tanıma bölgesini fare genomuna entegre edildi. Fare hücrelerinde meganükleaz sentezlendiğinde önce çift zincirli DNA kırıkları oluşturuldu. • Bu çift zincirli kırıklar verici bir homolog DNA kullanılarak ya homolog rekombinasyon (HR) ile yada hataya meyilli homolog olmayan uç birleştirme (error-prone non-homologous endjoining (NHEJ)) yöntemleri ile memeli hücrelerinin hedeflenmiş genetik modifikasyonu sağlandı. Böylece bu iki çalışmayla genom modifikasyonunun temellerini atılmış oldu. • Ancak meganükleazlar hücrelerde kromozomal DNAyı bölgeye özgü bir şekilde kesmelerine rağmen DNA tanıma bölgeleri oldukça uzun olduğundan, bu enzimleri genomik modifikasyon amacıyla tekrar programlanmaları (aa sekans değişikliği yaparak) oldukça güçtür. Bu sebeble gözler genomun istenen herhangi bir bölgesini modifikasyona uğratabilen programlanabilir ajanlara döndü. 3 Programlanabilir nükleazlarla genom mühendisliği • Programlanabilir nükleazlar; çift zincirli DNA kırıkları oluşturarak homolog rekombinasyon etkinliğini en az 100 kat arttıran yada hatalı homolog olmayan uç birleşimi sağlayarak (error-prone non-homologous end-joining (NHEJ)) hedeflendirilmiş mutasyon oluşumuna sebebiyet veren ajanlardır. • Birkaç yıl öncesine kadar zink finger nükleazlar (ZFNs) hedeflenmiş genom mühendisliğiyle ilgilenen araştırmacılar için tek seçenek olmasına rağmen 2011 yılının sonunda Nature Metod dergisi bu uygulamayı yılın uygulaması olarak seçtiğinde; ZFNlar transcription activator-like effector nucleases (TALENs)’ların yanı başında genom düzenleyici araçlar altında yer aldı. • Ocak 2013 yılında birkaç araştırmacı grubu birbirinden bağımsız olarak RNA-guided engineered nucleases (RGENs) ismi verilen yeni bir genom düzenleyici nükleaz sınıfının olduğunu duyurdular. • Bu üç nükleaz grubu da birbirinden farklı özellikleri olsa da benzer mekanizmalarla kromozomal DNA’yı bölgeye özgün bir ş ekilde kırıp, endojen DNA tamir mekanizmasını tetikleyerek hedeflenmiş genom modifikasyonuna yol açar. 4 Outcome of genome editing using programmable nucleases. Nuclease-induced strand breaks (DSBs) can lead to sequence insertion, nucleotide correction or change (red box) through homology-directed repair (HDR) in the presence of a donor DNA or a single-strand oligodeoxynucleotide (ssODN), both of which contain homology arms. DSBs can also be repaired through error-prone non-homologous end-joining (NHEJ), which does not require donor DNA or ssODN and consequently often leads to small insertions and deletions (indels). Typical indel sequences and the number of inserted (+3 and +1) or deleted (–2, –4 and –10) bases are shown. 5 Outcome of genome editing using programmable nucleases. b | When two DSBs are generated in cis on a single chromosome by programmable nucleases, the flanking region can be deleted or inverted. c | When two DSBs are generated on two different chromosomes, chromosomal translocations can be induced. 6 Genom mühendisliğinin kullanım yolakları. Genom mühendisliği yapısal manipülasyonla kromozomal yapıyı değiştiren, transkripsiyonel kontrolle gen fonksiyonunu ayarlayan, epigenetik değişimi tetikleyen ve DNA sekansını değiştirmek yoluyla da gen onarımı yapmaya yarayan metodolojik bir uygulamadır. 7 Zinc Finger Nükleazlarla genom mühendisliği • The Johns Hopkins Üniversitesinden Srinivasan Chandrasegaran isimli bir biyokimyacı; 1990 başlarında bir Tip IIS kesim enzimi olan FokI restriksiyon enziminin bir proteaz yardımıyla DNA’ya bağlanan bölgesinin ve DNA kesim nükleaz bölgelerinin birbirinden ayrılabildiğini gösterdi. Bu buluş Fok1 nükleaz bölgesinin bir DNA bağlanma bölgesine eklenerek sekans spesifik bir nükleaz proteini oluşturulabileceğini gösterdi. 1996 yılında Chandrasegaran ve arkadaşları Fok1 nükleaz bölgesine zinc finger proteinleri bağlayarak oluşturdukları ZFN ‘ların in vitro ortamda hedef DNA’yı spesifik olarak kırdıklarını gösterdiler. • Chandrasegaran’nın çalışmalarından esinlenen Dana Carroll ve arkadaşları Utah Üniversitesinde müşteri tasarımlı siparişle ZFN’ler oluşturarak ve sonrasında bunları Drosofila embriyolarına enjekte ederek ilk kez bir canlıda in vivo ortamda genom düzeltmeyi başardılar. Bundan sonraki çalışmalarda bitkilerde, hayvan ve insanlarda hedeflenen genler ZFN’lar kullanılarak başarıyla modifiye edildi. 8 ZFN’ların özeti A. ZFN’lar FokI kesim enziminin katalitik bölgesine 3 zinc finger proteini bağlayarak oluşturulur. Herbir zinc finger protein 3 nükleotide bağlanarak ZFN’ları genomda hedef bölgelere yönlendirir. Genomik DNA’nın birbirine karşı bölgelerinde iki ZFN hedeflenen DNA sekansıyla interaksiyona girerek DNA sekans özgünlüğü olmayan iki FokI nükleazın dimerize olmasını sağlar. B. FokI enzimlerinin dimerizasyonu hedef bölgede çift zincirli DNA kırığı (DSB) o l u ş t u r u r. H a t a l ı N H E J D N A t a m i r mekanizması gen sekansının bozulmasına sebebiyet verir. Alternatif olarak ortama DNA tamir dizisi konduğunda HDR aracılı genom modifikasyonuyla gen düzeltme veya ilavesi gerçekleştirilir. ZF = zinc finger protein. 9 Zinc Finger Nucleazların yapısı (ZFNs). Her bir ZFN amino ucunda bir zinc finger proteini ve karboksi ucunda da FokI nükleaz bölgesi içerir. X herhangi bir amino asidi temsil eder. ZFN çiftinin hedef sekansları ara bölgeler hariç yaklaşık 18-36 baz çifti uzunluğundadır. Zinc fingerlarin mutasyon yoluyla DNA tanıma özellikleri değiştirilerek programlanabilir nükleazlar oluşturulabilir. Hücre düzeyinde in vivo olarak ZFN aracılı başarılı genom düzenlemeleri yapıldıysa da ZFN’ların hedef özgünlüğü konusunda endişeler bir türlü giderilemedi. Modüler yöntemlerle önceden belirlenmiş Zinc Finger bölgeleri kullanılarak oluşturulan ZFN’lar hedef hücrelerde sekans benzerliği gösteren hedef dışı sekansları kesdiğinden dolayı sitotoksik bir etki gösterdi. Bu problemi düzeltmek için akademi ve endüstriden araştırmacılar ZFN’ların özgünlüğünü arttırabilmek için çalışmalar yaptılar. 10 TALEN genom modifikasyonu • ZFN’ların sitotoksizite problemine çözüm bir bakteriyel bitki patojeni olan Xanthomonas’dan 2009 yılında geldi. Biri Iowa State Üniversitesi’nde Adam Bogdanove’nin başını çektiği ve diğeri Martin Luther Üniversitesi’nde Ulla Bonas and Jens Boch isimli araştırıcı grubu Xanthomonas orijinli transkripsiyon aktivatör benzeri efektör proteinlerin (TALE) DNA ile interaksiyon mekanizmalarını açıklaması bu proteinlerin de genom modifikasyonun da kullanılabileceği düşüncesini doğurdu. • ZFN’lar ve TALEN’lar C-terminal uçlarında FokI nükleaz bölgesi olsa da DNA bağlanma bölgeleri birbirinden farklıdır. Zinc finger proteinlerinin aksine, TALE proteinlerinin her bir tekrarı tek bir bazı tanır. TALE proteinlerinin tekrar dizileri birbirleriyle değiştirilerek ve FokI bölgesine bağlanarak TALEN isimli DNA’ya bağlanan yeni bir nükleaz protein grubunu oluşturdu. 11 A. TALE proteininde bir N-terminal uç, merkezi DNA bağlanma bölgesi ve C-terminal ucunda da Nüklear lokalizasyon sinyali (NLS) ve aktivasyon bölgesi (AD) vardır. Her bir tekrar 34 aa uzunluğunda ve 12. ve 13. pozisyonlarında 2 çok değişken aa RVD (kırmızı renkli aa’ler) içerir. Her RVD bir spesifik DNA çiftini tanır ve herbir tekrar spesifik DNA sekansını tanıyarak aktivasyon bölgesi ile spesifik genlerin sentezini uyarır. B.TALEN proteininin yapısı. Timin (kırmızı), sitozin (yeşil), adenin (mavi) yada guanin (sarı) nükleotidlerine bağlanan 12 özgün tekrar içeren FokI nükleazla birleşik bir TALEN molekülü. Bir TALEN çiftinin hedef bölgeye karşılıklı bağlanması FokI dimerizasyonu sağlayarak çift zincirli DNA kırıkları (DSB) oluşturur. İstenilen genetik mutasyon NHEJ yada HDR yolağı ile sağlanır. 12 TALEN’ların yapısı. a | Transcription activator-like effector nuclease (TALEN) çiftinin şematik temsili resmi. Herbir TALEN amino ucunda transcription activator-like effector (TALEs) bölgeleri ve karboksil ucunda da FokI nuclease bölgesi vardır. 33-35 aa’den oluşan TALE tekrarları üzerlerindeki repeat variable diresidue (RVD; kırmızı) denen 12 ve 13 aa leriyle tek bir bazı tanır. TALEN çiftinin hedef sekansı ara bölgeler hariç yaklaşık 30-40 nükleoid çifti uzunluğundadır. 13 TALEN genom modifikasyonu • TALE tekrar dizilerinin olduğu DNA segmentinin oluşturulması oldukça zaman alıcıdır. Ayrıca Bu sekansların hücrelerde birbirleriyle rekombine olma ihtimali olduğundan bunları oluşturmak teknik açıdan oldukça güçtür. • TALEN’ların insan hücrelerinde minimal toksizite göstermesi oldukça dikkat çekici olsa da CRISPR-Cas9 sisteminin ortaya çıkması tüm ilginin bu yeni protein grubuna dönmesine yol açmıştır. 14 15 GENOME EDITING The new frontier of genome engineering with CRISPR-Cas9 Jennifer A. Doudna* and Emmanuelle Charpentier* BACKGROUND: Technologies for mak- ing and manipulating DNA have enabled advances in biology ever since the discovery of the DNA double helix. But introducing site-specific modifications in the genomes of cells and organisms remained elusive. Early approaches relied on the principle of site-specific recognition of DNA sequences by oligonucleotides, small molecules, or self-splicing introns. More recently, the site-directed zinc finger nucleases (ZFNs) and TAL effector nucleases (TALENs) using the principles of DNAprotein recognition were developed. However, difficulties of protein design, synthesis, and validation remained a barrier to widespread adoption of these engineered nucleases for routine use. ADVANCES: The field of biology is now ex- periencing a transformative phase with the advent of facile genome engineering in animals and plants using RNA-programmable CRISPR-Cas9. The CRISPR-Cas9 technology originates from type II CRISPR-Cas systems, which provide bacteria with adaptive immunity to viruses and plasmids. The CRISPRassociated protein Cas9 is an endonuclease that uses a guide sequence within an RNA duplex, tracrRNA:crRNA, to form base pairs with DNA target sequences, enabling Cas9 to introduce a site-specific double-strand break sgRNA binding to Cas9 Matching DNA target sequence CRISPR-Cas9 development DNA deletion DNA insertion DNA replacement DNA modification DNA labeling Transcription modulation RNA targeting ... PAM sequence CRISPR-Cas9 applications Biological research Research and development Human medicine Biotechnology Agriculture ... The Cas9 enzyme (blue) generates breaks in double-stranded DNA by using its two catalytic centers (blades) to cleave each strand of a DNA target site (gold) next to a PAM sequence (red) and matching the 20-nucleotide sequence (orange) of the single guide RNA (sgRNA). The sgRNA includes a dual-RNA sequence derived from CRISPR RNA (light green) and a separate transcript (tracrRNA, dark green) that binds and stabilizes the Cas9 protein. Cas9-sgRNA–mediated DNA cleavage produces a blunt double-stranded break that triggers repair enzymes to disrupt or replace DNA sequences at or near the cleavage site. Catalytically inactive forms of Cas9 can also be used for programmable regulation of transcription and visualization of genomic loci. SCIENCE sciencemag.org Corrected 25 November, 2014; see full text. Published by AAAS OUTLOOK: CRISPR-Cas9 has triggered a revolution in which laboratories around the world are using the technology for innovative applications in biology. This Review illustrates the power of the technology to systematically analyze gene functions in mammalian cells, study genomic rearrangements and the progression of cancers or other diseases, and potentially correct genetic mutations responsible for inherited disorders. CRISPR-Cas9 is having a major impact on functional ON OUR WEB SITE genomics conducted in experimental sysRead the full article tems. Its application in at http://dx.doi .org/10.1126/ genome-wide studies science.1258096 will enable large-scale screening for drug targets and other phenotypes and will facilitate the generation of engineered animal models that will benefit pharmacological studies and the understanding of human diseases. CRISPR-Cas9 applications in plants and fungi also promise to change the pace and course of agricultural research. Future research directions to improve the technology will include engineering or identifying smaller Cas9 variants with distinct specificity that may be more amenable to delivery in human cells. Understanding the homology-directed repair mechanisms that follow Cas9-mediated DNA cleavage will enhance insertion of new or corrected sequences into genomes. The development of specific methods for efficient and safe delivery of Cas9 and its guide RNAs to cells and tissues will also be critical for applications of the technology in human gene therapy. Downloaded from http://science.sciencemag.org/ on December 18, 2016 REVIEW SUMMARY in the DNA. The dual tracrRNA:crRNA was engineered as a single guide RNA (sgRNA) that retains two critical features: a sequence at the 5 side that determines the DNA target site by Watson-Crick base-pairing and a duplex RNA structure at the 3 side that binds to Cas9. This finding created a simple two-component system in which changes in the guide sequence of the sgRNA program Cas9 to target any DNA sequence of interest. The simplicity of CRISPR-Cas9 programming, together with a unique DNA cleaving mechanism, the capacity for multiplexed target recognition, and the existence of many natural type II CRISPR-Cas system variants, has enabled remarkable developments using this cost-effective and easy-to-use technology to precisely and efficiently target, edit, modify, regulate, and mark genomic loci of a wide array of cells and organisms. The list of author affiliations is available in the full article online. *Corresponding author. E-mail: doudna@berkeley.edu (J.A.D.); emmanuelle.charpentier@helmholtz-hzi.de (E.C.) Cite this article as J. A. Doudna, E. Charpentier, Science 346, 1258096 (2014). DOI: 10.1126/science.1258096 28 NOVEMBER 2014 • VOL 346 ISSUE 6213 1077 16 RNA-öncülü modifiye nükleazlar RGEN • CRISPR/Cas proteinleri, bakteri ve arkealarda virus ve patojenlere karşı korunma amacıyla CRISPR RNA (crRNA)-tabanlı DNA tanıma ve Cas nükleaz-aracılı DNA kesim yoluyla bağışıklık sisteminde görev alan proteinler olarak keşfedilmişlerdir. CRISPR/Cas sistemi sekansı yapılan bakterilerin %40’ında arkeaların ise %90’ında tesbit edilmiştir. • Bakteriler kendi hücresine invaze olan fajlar yada plazmidlerden küçük DNA parçalarını (20 baz uzunluğunda) yakalayıp bunları kendi genomlarına integre ederek CRİSPR genom lokusu oluştururlar. • Tip 2 CRISPR sisteminde bu CRISPR bölgeleri pre-CRISPR RNA (precrRNA) olarak transkripte olur, işlendikten sonra da hedef spesifik crRNA oluşturur. pre-crRNA’nın işlenmesine aynı bölgeden transkripte olan transactivating crRNA (tracrRNA)’da katkı sağlar. 17 CRISPR/Cas bacteriyel bağışıklık sisteminin özeti. (A) CRISPR lokusunun tipik yapısı; (B) Yeni spacer DNA kazanım ve invaze DNA kırılımının şematik görüntüsü. CRISPR lokusu spacer (aralık) adı verilen birbirinden farklı tekrarlanmayan (tekrar dizisi olmayan) aralıklı lokalize bir dizi korunmuş genomik sekansdan oluşur. CRISPR/Cas sisteminde, hücreye invazyon yapan yabancı DNA, Cas nükleaz tarafından işlenerek küçük parçalara bölünür ve spacer DNA olarak CRISPR lokusuna integre edilir. Bu spacerlar virus ve faj enfeksiyonlarına bir cevab olarak crRNA üretiminde transkripsiyonel template olarak virus ve fajların hedef sekansını Cas aracılı kırılımı için kullanılır. 18 CRISPR/Cas9-aracılı DNA kesimi. İşlenmiş crRNA Cas9 proteinini invaze faj DNA’sının hedef bölgesine yönlendirir. Cas9 bir zincirde RuvC-nükleaz bölgesiyle, diğer zincirde ise (crRNA komplementer bölge) HNH nükleaz bölgesiyle hedef sekansda çift zincirli kırık oluşturulur. CRISPR/Cas9-aracılı DNA kesimi (özgünlüğü) hedef sekansda crRNA'ya komplementer bir dizi ve hedef sekansın hemen aşağısında da 3 nükleotid PAM sekansı gerektirir. 19 CRISPR/CAS9-aracılı genom modifikasyonu • Hem crRNA hem de tracrRNA, CRISPR-associated protein 9 (Cas9) ile komplex oluşturarak dualRNA–Cas9 denilen aktif DNA endonükleaz oluşturur. RNA yüklemesi Cas9 proteininde konformasyonel değişim yaparak hedef DNA’nın içeri girmesini sağlayan bir kanal oluşturur. • Oluşan endonükleaz; 20 nükleotid dizisi crRNA’ya komplementer olan (protospacer) ve 3 dizisi de protospacer bitişik motif (PAM) olarak bilinen (5ʹ-NGG-3ʹ) özellikle Cas9 proteini tarafından tanınan 23 baz çifti uzunluğunda hedef DNA sekansını kırar. • crRNA ve tracrRNA RGEN’leri basitleştirmek amacıyla tek zincirli öncül RNA (sgRNA) oluşturmak amacıyla birbirine bağlanabilir. 20 CRISPR/CAS9-aracılı genom modifikasyonu • CRISPR/Cas sistemi Cas proteinin yapısı ve sekansına bakıldığında tip 1, tip 2 ve tip 3 olmak üzere sınıflandırılabilir. Tip 2 CRISPR/Cas sisteminde HNH nucleaz bölgesi ve RuvC-benzeri nücleaz bölgesi içeren yalnızca tek bir Cas proteinine ihtiyacı vardır. • Spesifik DNA bölgelerini kesebilmek için in vitro ortamda crRNA ve tracrRNA‘nın birleştirilerek tek bir öncül RNA oluşturulması (guide RNA sgRNA); Cas9 aracılı programlanmayı daha da basitleştiren bu alandaki en önemli buluşlardandı. • CRISPR/Cas9-aracılı genom düzeltmesinde, sgRNA hedef sekansı (protospacer) komplementer baz eşleşmesiyle tanır ve bağlanır. 21 CRISPR/Cas sisteminin çalışma Mekanizması. A. Yabancı DNA sekansının parçalanması. Hücrelere virus veya plazmidler girdiğinde c r R N A l a r y a b a n c ı D N A n ı n PA M d i z i l i protospacer sekansına bağlanır. TracrRNA crRNAnın komplementer DNA sekansına bağlanmasını ve Cas9 nükleazla interaksiyona girmesini kolaylaştırarak, crRNA bağımlı çift zincirli DNA kırığı (DSB) oluşturur. Bölgeye özgü çift zincirli DNA kırığı PAM sekansının hemen 3 baz çifti önünde (siyah oklar) gerçekleşir. B.CRISPR/Cas sisteminde genom modifikasyonu. Tasarımı yapılmış gRNA (crRNA ve tracrRNA kimerası-hibridi) genomik DNA’da PAM bitişik hedef sekansı tanıyarak Cas9 aktivasyonu sağlayıp hedef spesifik DSB oluşturur. Akabinde genom düzeltme için DNA tamir mekanizması devreye sokulabilir. 22 CRISPR/Cas9 systeminin çalışma mekanizması. Tek bir guide RNA (sgRNA) konakçı hücrede komplementer baz eşleşmesiyle hedef sekansı (protospacer) tanır. Cas9 nükleaz Protospacer Adjacent Motif (PAM) ‘in yanında (3’ pozisyonunda sekansı NGG olan) çift zincirli DNA kırığı oluşturur. 23 RGEN lerin yapısı. Resimde bir RNA-guided engineered nuclease (RGENs) ş ematize edilmiştir. RGEN bir Cas9 ve tek zincirli öncül RNA (a single-chain guide RNA (sgRNA)) gerektirir. crRNA daki öncül sekans (sgRNA) protospacer adjacent motifin (PAM sekansı, 5ʹ-NGG-3ʹ) yanında protospacer olarak bilinen 20 baz çiftine komplementerdir. c | RGEN tarafından kırılıp hedef DNA da küt uç oluşturması gösterilmiştir. 24 CRISPR–Cas9 ile RNA bağımlı genom modifikasyonu • ZFN ve TALEN sisteminde FokI nükleazların yalnızca bu enzim dimerize olduğunda hedef DNA yı kırması bu proteinlerin hedef özgünlüğünü arttıran bir durumdur. Bunun tersine Cas9 monomerik formda aktifdir. Dahası CRISPR–Cas9 prokaryot orijinli olup bakteriofajlar gibi genetiği hızla değişebilen infeksiyonlara karşı kazanılmış bağışıklık sisteminde fonksiyon görür. Bu sebeble Cas9 proteinlerinin düşük DNA sekans özgünlüğü farklı DNA sekanslarını tanımak ve kırabilmek için gerekir. • ZFN ve TALEN’ların aksine Cas9 proteininin yüksek ökaryötik hücrelerde bilinen fonksiyonlarının olmaması ve ökaryötik hücrelerin genomun devasa büyüklüğü Cas9 nükleazların düşük sekans özgünlüğünün bu hücrelerde hedef dışı dizileri kırabileceği endişesini doğurdu. Bu nedenle, araştırmacılar Cas9 proteininin genom üzerindeki muhtemel hedef bölgelerin belirlenmesi ve bu proteinin özgünlüğünü arttıracak yöntemler üzerine yoğunlaştılar. 25 CRISPR–Cas9 sisteminde hedef dışı (offtargeting) etkileri gidermenin yolları • Bu amaçla çift zincirli DNA kırıkları oluşturan nükleazların yerine tek zincirli DNA kırıkları oluşturan (SSB) nikazların kullanılması önerilmiştir. 2 aktif bölgeli Cas9 yerine aktif bölgedeki anahtar bir aminoasidin (örneğin D10) alanine dönüştürülmesiyle (D10A Cas9) DNA nikazlar elde edilir. SSB hatalı NHEJ ile tamir edilmese de HR ile tamir edilebilir bu da istenilen genom modifikasyonunu sağlayabilir. Ancak SSB tamir etkinliği DSB tamirinden 10 kat daha az olması genom modifikasyonunda nikazların kullanımını sınırlandırmaktadır. • Bu sınırlandırmayı aşabilmek için her biri birbirine yakın tek bir DNA zinciri kıran bir çift DNA nikaz kullanarak genom modifikasyonunun hem etkinliği hem de özgünlüğü arttırılarak DSB oluşturulabilir. Hedef dışı SSB endojen olarak SSB tamir enzimleriyle başarılı bir ş ekilde tamir edildiğinden Cas9 nükleazlar tarafından oluşturulan hedef dışı mutasyonlar Cas9 nikazlar tarafından kolay kolay oluşturulmazlar. Daha da önemlisi bu gelişme programlanabilir nükleazların gen tedavisinde kullanımını sağlamıştır. 26 27 Programlanabilir nükleazlarla gensiz gen tedavisi • Barbas ve grubu 2012 yılında saflaştırılmış rekombinant ZFN’ların hücrelere penetre olabilen özelliklerinin olduğunu keşfetmiş ve bu ZFN proteinlerin direk olarak hücrelere verilmesinin (ZFN proteinlerinin yarı ömrü kısa olduğundan) genomda uygulamanın hedef dışı etkileri (off targeting) azalttığını bulmuşlardır. • Bu çalışmadan esinlenen diğer araştırmacılar rekombinant Cas9 guide-RNA ribonükleoproteinleri (RNP) olduğu gibi hayvan embriyolarına, bitkilere ve memeli hücrelerine aktarmışlardır. Cas9 RNPlerin hücrelerde çabucak parçalanması bu proteinlerin olası hedef dışı etkilerinin azalmasını sağlamıştır. • Cas9 RNPler hücrelere mikroenjeksiyon, elektroporasyon, lipofeksiyon ve protein transdüksiyonu gibi farklı yöntemlerle verilebilir. Burada genlerin plazmid (transfeksiyon) veya viral vektörlerle aktarılmasını içeren konvansiyonel gen tedavi denemelerinden farklı olarak Cas9 RNP naklinde eksojen DNA aktarımı olmaz. • Dolayısıyla yabancı DNA’ya karşı olası bir bağışıklık yanıtının gelişmesi yada yabancı DNA’nın konakçı genomunda istenmeyen bir bölgeye entegrasyonu söz konusu değildir. 28 29 30 CRISPR/CAS9-aracılı genom modifikasyonu • PAM sekanslı herhangi bir genomik lokusu hedefleyebilmek için sgRNA’daki 20 nükleotid uzunluğundaki öncül sekansı değiştirerek nückleazlar tekrar programlanabilir. Hatta birden fazla sgRNA kullanarak aynı anda birden fazla genomik lokusun değiştirilmesi (multiplexing) mümkündür. • Diğer tasarımı yapılmış nükleazlar gibi Cas9 hedef bölgede çift zincirli DNA kırıkları oluşturarak (DSBs) endojen DNA tamir mekanizmalarını tetikler. Homolog bir verici DNA varlığında homology-directed repair (HDR) mekanizmasıyla DNA tamiri gerçekleştirilir. Homolog verici DNA yokluğunda da nonhomologous end-joining (NHEJ) mekanizmasıyla tamir mekanizmasının doğal bir sonucu olarak rekombinasyon bölgesinde inzersiyonlar, delesyonlar (indel) yani mutasyonlar oluşur. • Bu sebeble, eksojen HDR ş aplanları oluşturularak Cas9 ve sgRNA varliğında hücrelere verildiğinde genomda istenen sekans değişimi gerçekleştirilir. Ancak bu işlem yalnızca bölünen hücrelerde düşük miktarda gerçekleşir. 31 32 33 Cas9 proteinin fonksiyonel bölgelerinde modifikasyon ve sonuçları • A: The Cas9 nükleaz hedef DNA’nın komplementer veya komplementer olmayan DNA zincirini HNH ve RuvC bölgeleriyle kırar. • B: RuvC bölgesinde D10A mutasyonu olduğunda Cas9 nikaz olarak fonksiyon görür. • C: HNH bölgesinde H840A mutasyonu olduğunda da Cas9 yine nikaz olarak fonksiyon görür. • D: Hem RuvC hem de HNH bölgelerinde mutasyon taşıyan Cas9 proteini (dead dCas9) yalnızca RNA tarafından yönlendirilmiş DNA’ya bağlanan bir protein olarak fonksiyon görür. • E: dCas9 proteinine bir aktivatör veya baskılayıcı bölgeler ekleyerek gen ekspresyonu aktive edilebilir veya susturulabilir. • F: Schematic representation of double 34 nicking. 35 CRISPR/Cas9 sistemiyle yeni tasarımlar ve deneysel yaklaşımlar • Günümüzde Cas9 proteinin farklı formları olup bunlarla genomde çok farklı değişiklikler yapılabilmektedir. Örnekler: • a) Doğal (değiştirilmemiş-orijinal) Cas9’la çift zincirli DNA kırıkları oluşturmak, • b) Cas9 nikazlarla tek zincirli DNA kırıkları oluşturmak. Bu durumda farklı DNA zincirini hedefleyen birbirine yakın 2 gRNA kullanarak çift zincirli DNA kırığı oluşturmak mümkün, • c) Nukleaz-yoksun Cas9 (dCas9) kullanarak CRISPR’la gen aktivasyonu. dCas9 proteinine VP64 gibi transkripsiyon aktivasyon bölgeleri eklenerek hedef genler aktive edilebilir. • d) Bunun tersine dCas9 proteinine transkripsiyonu baskılayan bölgeler eklenerek (dCas9KRAB) hedef genler geçici olarak susturulabilir. • e) Donor plazmidlerde floresan kodlayan genler ve hedef bölgeye homoloji gösteren sekans varlığında arzu edilen protein genleri floresan kodlayan dizilerle işaretlenebilir. • f) Cas9-WT yada Cas9 nikaz kullanarak HDR mekanizmasıyla hedef genlerde nokta mutasyonları oluşturulabilir. 36 37 38 Özgünlük ve etkinlik • Hedefli genom modifikasyonunda en önemli sorunlardan biri hedef özgünlüğüdür. İstenen, genomun diğer bölgelerine dokunmadan yalnızca hedef bölgede DNA sekans değişikliğidir. • TALEN ve CRISPR-Cas9 sistemi yaygın olarak kullanılmalarına rağmen herkisinin de hedefleme açısından mükemmel özgünlük sağladığı söylenemez. Başka bir deyişle bu nükleazların genomun diğer yerlerinde de değişiklik yaptığı saptanmıştır. • Bu hedef dışı etkileri hücrelerde, organlarda, organizmada hatta çevrelerinde istenmeyen yan etkiler oluşturabilir. Bu sebeble bu nükleazların genom modifikasyonlarında kullanılmasını sınırlayan en önemli sebeb hedef özgünlüğüdür. 39 40 41 42 Hedef dışı bölgeleri saptamada ilk jenerasyon metodlar: • Hedef dışı sekanslara karşı modifiye edilmiş nükleazların (engineered nucleases-EN) özgünlüğünü test etmek için kullanılan metodlardan biri SELEX (systematic evolution of ligands by exponential enrichment) yöntemidir. Bu yöntemde modifiye edilmiş nükleazlar oluşturulan DNA kütüphanesiyle birlikte karıştırılır, nükleazların bağlandığı DNA fragmentleri seçilir ve çoğaltılır. Elde edilen DNA fragmentlerine DNA adaptörleri eklenerek gelecek jenerasyon sekanslamaya (NGS) tabi tutulur. • Bu yöntemde modifiye edilmiş nükleazlar normalde uzun sekansları tanıdığından içinde her muhtemel nükleotid kombinasyonu barındıran büyük bir çift zincirli DNA kütüphanesi oluşturulmasını gerekiyor. Örneğin 18 sekanslı bir hedef DNA dizisinde tarama için 4e18 (7 × 10e10) DNA fragmenti içeren bir DNA kütüphanesi oluşturuldu. Ancak bu tip büyük kütüphanelerin hem üretimi hem de herkes tarafından kullanımı pratik ve yaygınlık açısından mümkün olamadı. • Bu ilk jenerasyon tarama metodları yalnızca sınırlı sayıda potansiyel hedef dışı bölgeleri tarayabildiğinden genomun tamamı yeterince tarandığında bulunandan çok daha fazla sayıda hedef dışı değişimlerin olabileceği endişesi gündeme geldi. 43 2. jenerasyon metodlar: Nükleaz inaktif dCas9’un bağlanma özgünlüğünü belirlemede genom çaplı tarama yöntemleri • Chromatin immunoprecipitation sequencing (ChIP-seq). Özgün ve inaktif modifiye nükleazlar (EN’ler) hücrelere transfekte edilir. DNA’ya bağlanan tüm proteinler birbirlerini bağlayan (kroslinking) ajanlarla in vivo olarak fikse edilir. İzole edilen genomik DNA sonrasında küçük parçalara bölünür. EN’lere bağlanan DNA parçacıkları EN’lere karşı geliştirilen antikorlarla çöktürülerek (immunopresipitasyon) gelecek jenerasyon sekanslamaya tabi tutulur. • Bu teknik farklı gRNA’larla kompleks dCas9’un genom çapında bağlanabileceği bölgeleri taramak için kullanıldı. Sonuçlara göre bazı gRNA’lar spesifik bölgelerle interaksiyona girse de bazı gRNA’ların spesifik olmayan 1200 kadar farklı bölgeyle interaksiyona girdiği tesbit edildi. 44 3. Jenerasyon metodlar: Cas9 tarafından oluşturulan çift zincirli DNA kırıklarının genom çaplı taraması • Pek çok canlıda çift zincirli DNA kırıkları (Double strand breaks (DSB)) DNA kırık noktasında mutasyon oluşmasına sebebiyet veren hataya meyilli homolog olmayan uç birleştirme yolağıyla (error-prone non-homologous end-joining pathway) tamir edilir. • Bu açıdan katalitik olarak aktif Cas9 proteinin genomun herhangi bir tarafında (genom çapında) sebebiyet verdiği çift zincirli DNA kırıkları tüm genom sekanslamayla (whole genome sequencing (WGS)) en doğru şekilde bulunabilir. • Ancak WGS, genomda gözlemlenen yüksek frekanslı değişimleri kolaylıkla saptasa da, bu teknikle tüm genomu 30-60 kez sekanslamak düşük frekanslı mutasyonları tesbit etmek için yeterli değildir. Dolayısıyla WGS tekniğinde yoğun sekans derinliği (extensive sequencing depth) bu tekniğin etkinliğini sınırlayan bir faktördür. • Hele hele insan kökenli olmayan hücrelerin bu yöntemle taranmasında sekans derinliğine ilaveten maliyet de sınırlayıcı bir faktör olarak karşımıza çıkmaktadır. 45 3. Jenerasyon metodlar: • İntegraz özürlü lentivirus vektörleri (Integrase-deficient lentivirus vectors IDLV) Cas9 tarafından oluşturulan çift zincirli DNA kırıklarına bir belirteç gen yerleştirerek genomda hedef dışı bölgeleri tesbit etmede kullanıldı. Önce modifiye nükleazlar (Engineered nucleases EN) hücrelere transfekte edilir sonra da puromisin taşıyan İDLV’lerle hücreler transdüksiyona uğratılır. DSB’lerin NHEJ yolağıyla tamiri üzerinde puromisin dirençlilik geni taşıyan IDLV partiküllerin genoma integrasyonuna sebebiyet verir. • Puromisin dirençli hücrelerden genomik DNA izole edilir. IDLV üzerinde mevcut LTR (long terminal repeat)’lere karşı geliştirilen primerler kullanılarak linear amplification– mediated PCR’ la (LAM-PCR) IDLV integrasyon bölgeleri tesbit edilir. Adaptör barkodların ilavesiyle PCR ürünü gelecek jenerasyon sekanslama tekniğiyle (NGS) 46 analiz edilir. 3. Jenerasyon metodlar: • The GUIDE-seq approach (genome-wide, unbiased identification of DSBs enabled by sequencing) GUIDE-seq tekniği de benzer ş ekilde çift zincirli DNA kırıklarına küçük bir barkot ekleyerek ve bunları yüksek çıktılı sekanslamaya (high throughput sequencing) tabi tutarak genom üzerinde hedef dışı bölgelerin belirlenmesini sağladı. • Bu teknikde modifiye nükleazlar özgün bir çift zincirli oligodeoksinükleotidle (dsODN) birlikte hücrelere transfekte edilir. Çift zincirli DNA kırıkları hataya meyilli homolog olmayan uç birleştirme yolağıyla (error-prone non-homologous end-joining pathway) tamir edildiğinde dsODN lar genoma integre olur. Genomik DNA transfekte hücrelerden izole edilip parçalanır. Sekans adaptörleri ilave edilerek NGS yöntemiyle integrasyon bölgeleri (çift zincirli DNA kırıkları) tesbit edilir. • GUIDE-seq tekniği hedef dışı çift zincirli kırık olasılığının gRNA ya bağlı olarak 0 ile 150 arasında değiştiğini gösterdi. 47 • Digenome-seq. Saflaştırılmış genomik DNA modifiye endonükleazlarla in vitro ortamda inkube edilir. Genom parçalanıp gelecek jenerasyon sekanslamayla analiz edilir. • Digenome-seq tekniğinin programlanabilir nükleazların hedef dışı etkilerini belirleyebilmek için kullanılabilecek güçlü, duyarlı, ve maliyeti düşük bir teknik olduğu ileri sürülüyor. 48 Sonuç: CRISPR/Cas9 ne kadar özgün? • Bu çalışmaların hepsinden çıkan ortak sonuç, CRISPR/Cas9 aracılı kırıklar; tasarımı yapılan sgRNA’a bağlı olarak yüksek veya düşük özgünlükte olabilir. Dolayısıyla, CRISPR/Cas9 sisteminde özgünlük tamamıyla sgRNAların spesifisitesine bağlanabilir. Bazı sgRNAlar çok özgünken bazıları sekans özgünlüğü yetersizliği sebebiyle yüzlerce hedef dışı sekanslara kolaylıkla bağlanabilir. • Şu anda araştırmacılar hangi sgRNAların en az hedef dışı etki sergilerden hangilerinin en fazla hedef dışı etki gösterebileceğini belirlemeye çalışıyorlar. • Bu bakımdan en azından CRISPR/Cas9 sisteminin ZFN ve TALEN’lara oranla teknolojik olarak daha önde ve kullanımlarının daha yaygın olduğunu belirtmekte yarar var. 49 Therapeutic genome editing using CRISPR-Cas9 50 51 52 Modifiye nükleazların geleceği • Bir zamanların gizemli prokaryotik viral savunma sisteminin günümüzde canlıların genetik yapısını değiştirebilmek için kullanılabilecek en etkili araç olması tıbbi araştırmalarda temel bilimlerinin rolünün açığa çıkması açısından önemlidir. Faj ve bakteriler arasındaki savaşta temel restriksiyon enzim araştırmaları nasıl rekombinant DNA teknolojisini geliştirdiyse, bugünün Cas9 temelli genom modifikasyon araçları da mikrobiyal antifaj savunma sisteminin bir parçası olarak ortaya çıkıp genom mühendisliğinin en etkin yöntemleri haline geldi. • Birkaç yıl öncesine kadar genom modifikasyonu dünyanın farklı köşelerinde sınırlı sayıda laboratuvarlarda yapılan bir işlem olarak bilinirken, bugün için biyomedikal araştırmalarda yaygın olarak herkes tarafından kolayca kullanılabilecek bir metod seviyesine çıktı. • Genom mühendisliği; kişilerde tıp ve biyoteknolojik araştırma açısından heyacan yaratsa da kitlelerde de bir endişe yarattığı da bir gerçek. Gelecekte CRISPR–Cas9 kullanılarak insan, hayvan ve bitkilerin genom modifikasyonu yoluyla en azından hastalıklara yatkınlık gibi istenmeyen özelliklerin giderildiği istenilen özelliklerin de kalıtıldığı bir dünyada yaşamak durumunda kalabiliriz. • Şu an için modifiye edilmiş nükleazların klinik denemelerde olduğunu sonuçlarını görebilmemiz için de biraz beklememiz gerektiğini söyleyebiliriz. 53 İlgiyle dinlediğiniz için teşekkürler Prof. Dr. Salih Sanlioglu Akdeniz Üniversitesi Gen ve Hücre Tedavi Merkezi, Antalya, Türkiye, 07058 54